

(中国器审 2024年08月15日)
单分子测序(Single-molecule Sequencing,SMS)是在第一代Sanger测序、第二代NGS高通量测序技术的基础上发展起来的第三代测序技术。因其测序时DNA分子无需PCR扩增,实现了对每一条DNA分子的单独测序而得名。Helico Bioscices公司基于合成测序理论于2008年推出了世界上第一款单分子测序平台HeliScope,但其测序的平均读长相对较短,系统整体测序错误率较高[1]。之后出现了单分子的长读长测序技术,目前已经实现商业化的长读长测序技术主要有Pacific Biosciences(PacBio)公司的单分子实时测序技术(Single-molecule Real-time,SMRT)和Oxford Nanopore公司(Oxford Nanopore Technologies,ONT)的纳米孔测序技术[2]。SMS和SMRT技术主要是将4种不同的碱基转化为荧光信号,然后通过转化放大后的信号对碱基进行区分。而纳米孔测序技术则是将4种碱基转换为电信号,然后对电信号进行收集、整理、转化与数据输出,与单分子荧光测序技术在信号处理上存在较大差异,故而纳米孔测序技术也被称为第四代测序技术。本文将主要介绍纳米孔单分子测序技术的原理、特点及其在体外诊断领域的应用。
一、纳米孔测序的原理
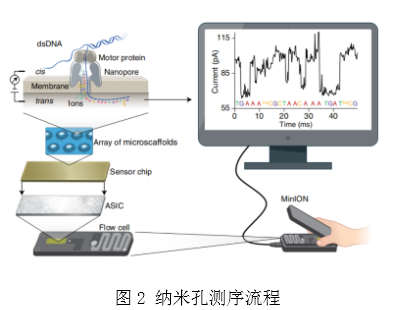
纳米孔单分子测序技术是基于电信号测序的技术,相对于其他测序技术, 纳米孔测序技术的样本处理极其简单,无需DNA聚合酶或者连接酶,也无需dNTPs,其测序过程中包含了以下几种关键物质,如图1、图2所示。

纳米孔蛋白(Nanopore),可以嵌入到细胞膜中作为离子或分子通道的跨膜蛋白,具有天然的蛋白纳米孔。
多聚物薄膜(Membrane),跨膜蛋白会被嵌入到高电阻率的由人工合成的多聚物膜中,膜两侧是离子溶液,在两侧加不同的电位,离子就会在孔中流动,形成电流。
马达蛋白(Motor protein),在纳米孔测序文库构建时,需要在接头上连接一种马达蛋白,用于将DNA或RNA分子推入纳米孔中。
连接臂(Tether),用于锚定DNA或RNA链,防止其在溶液中飘动,并使其进入纳米孔中。
将人工合成的多聚物膜浸没在离子溶液中,膜上布满了由解旋酶和蛋白孔两部分组成的跨膜通道蛋白纳米孔,在膜两侧施加不同电压形成电压差。由于多聚物膜不可导电,电流只能通过纳米孔进行传导。连接臂引导待测链进入纳米孔,待测链在马达蛋白的牵引下经解螺旋后以单链形式穿过纳米孔(如图1)。不同碱基在通过纳米孔的恒定的电场时会引起电流不同幅度的变化,使用计算机软件及人工智能算法识别并推断出碱基类型,从而完成了DNA或RNA的测序[3](如图2)。
二、纳米孔的类型
1.生物纳米孔
纳米孔测序的概念首次在80年代被提出,并且随着纳米孔蛋白及马达蛋白的技术发展而得以实现。第一个被发现可以通过DNA或RNA影响离子电流并能使之被检测到的纳米孔蛋白是1996年Kasianowicz等提出的α-溶血素蛋白[4]。但是因DNA通过纳米孔的速率过快,而无法获得有效的电流信号。之后的2010年,耻垢分枝杆菌蛋白A(Msp-A)纳米孔被发现可以减缓DNA穿过纳米孔的速度,提高DNA单碱基的检测灵敏度[5]。随后,2014年研究者发现使用phi29 DNA聚合酶作为马达蛋白[6],可以控制DNA穿过纳米孔的速度。
生物纳米孔主要包含以上3种,均为由某种蛋白质分子镶嵌在磷脂膜上形成的天然纳米孔,可以进行灵活的生物化学修饰。然而生物纳米孔在膜稳定性、 电流噪声等方面的问题在一定程度上限制了其发展。牛津纳米孔公司在蛋白纳米孔的应用方面取得了一定进展, 他们的GridION和MinION系统就是基于生物蛋白纳米孔的测序平台。
2. 固态纳米孔
Li等在2001年开启了固态纳米孔的研究[7]。固态纳米孔主要是在氧化硅、石墨烯等固态材质上通过离子束刻蚀等技术制备出的纳米孔,因其尺寸可调、可靠性高、易修改等优点,被广泛应用于DNA测序、蛋白质检测和能量转换等研究领域[8],其中比较常见用于DNA检测的固态纳米孔是氮化硅纳米孔和石墨烯纳米孔。
相比于生物纳米孔,固态纳米孔在稳定性、电流噪声、工艺集成方面有着显着的优势,但是因为受限于如今的半导体工艺制造水平,固态纳米孔的制造还较为复杂与昂贵。
三、纳米孔测序的特点
相比于其他测序平台,纳米孔测序作为一种新型测序技术在成本、速度、读长和准确率等诸多方面有着不同的特点,其优势十分显著,主要可以概括为以下几点:
1.较长的测序读长
纳米孔测序技术利用碱基穿过纳米孔时电信号的改变实现测序,理论上可检测通过纳米孔的全部核酸序列,读长长度仅受限于所测单链DNA的长度[9]。长度长可以提供更完整、更连续的基因组组装,在具有大型结构变异和高水平重复区域的基因组中优势显著。
2.可快速实时测序
相比于其他传统测序,纳米孔测序真正意义上做到了动态实时测序,可边测序边输出结果,用户可以在测序早期了解样本的质量和状态,也可以在获得足够的数据后停止测序。同时纳米孔测序技术所需的时长也是远远短于其他测序方法,节约了操作时间同时降低了测序成本。
3.纳米孔测序设备简单便携
目前使用最成熟的MinION测序仪尺寸只有一支笔的长度,重量大约100g,长度仅10cm,可使用高速USB插入电脑,以其极小的体积彻底颠覆了人们对测序仪的印象,被称为“U盘测序仪”[10]。因其便携性,可在实验室、野外甚至太空等各种复杂环境下完成实时测序,可保证对突发疫情处理的时效性。
4.可直接对RNA进行测序
纳米孔测序可以直接对各类原始DNA和RNA进行测序,不仅节省了时间和成本,完整地保留碱基修饰的信息,还能避免将RNA逆转录为DNA扩增所产生的偏向性及可能引入的突变。其文库制备的工作流程也较为简单。
虽然纳米孔测序的优点十分明显,与前几代技术相比在成本、速度方面有着很大优势,但是目前还处在起步阶段,从测序原理到制造工艺都存在有许多问题。首先,检测准确度相对较低,这是纳米孔测序发展过程中一直致力于解决的问题,除了通过优化纳米孔和马达蛋白外,还通过独立研究开发的算法来解决准确度的问题。其次,检测通量和文库产量也限制了该技术的应用。目前仍然缺乏针对少量样品的高产量文库制备和测序规范流程。而且也并非总是能够从临床样本中获得足够大且完整的高分子量DNA和全长RNA。读取长度和产量之间仍需权衡。
四、纳米孔测序的应用
纳米孔测序有独特的优势:长度长,测序速度快,这让它在某些场景中占有很大的优势,但是通量偏低、价格高、准确度偏低也严重限制了它的应用场景。根据之前的分析,对读长要求高,但是对序列数和准确度要求不高的应用场景才是纳米孔测序的最适场景。
1.大基因组拼接
在以往基于短片段的基因组拼接中,由于一些动植物基因组本身具有多倍体,高度重复,高度杂合的特性,导致基因组拼接异常艰难。而纳米孔测序技术具有长读长的特点,利于大基因组的拼接,可以极大的提高基因组的完整性。
2.全长转录组
以往的转录组分析由于无法直接对RNA进行测序,往往需要先对mRNA进行打断,再反转录为cDNA,无法获取和分析全长转录本。纳米孔的长读长特点可以准确识别各基因的多个同源异构体,简单准确;并且可以直接测序RNA,直接识别RNA的碱基修饰。
3.大片段结构变异
基因组上会产生很多与人类疾病相关的大片段结构变异(如缺失、倒位和易位等),短测序读长无法准确检测这些变异,而纳米孔测序的读长较长,适合进行大片段结构变异的检测,在疾病研究等方面具有良好的发展前景。
4.病原微生物快速鉴定[11]
由于纳米孔测序具有实时,快速的特点,可以在采集点直接进行测序,实时得到序列信息进行物种分类鉴定,完成病原微生物的快速鉴定。目前纳米孔测序技术在传染病、临床感染病原快速检测中的应用研究较为广泛。
五、结论与展望
纳米孔测序技术近年来发展迅猛,相比于其他测序技术,其凭借着超长读长、实时监测、简单便携等特点开始被广泛应用于各个领域,但是目前纳米孔测序技术也不尽完美。目前国内外多家企业在研发准备申报相关单分子测序产品的过程中,主要集中在病原体检测和肿瘤、遗传病早筛等方向,后续我们会继续关注相关产品的准确度、检测通量及文库制备的相关技术进展。
参考文献:
[1] Steinmann KE,Hart CE,Thompson JF,Milos PM. Helicos Single-molecule Sequencing of Bacterial Genomes[J]. High-Throughput Next Generation Sequencing,2011,733:3–24.
[2] Maitra RD,Kim J,Dunbar WB. Recent Advances in Nano-pore Sequencing[J]. Electrophoresis,2012,33 (23):3418–3428.
[3] Wang Y, Zhao Y, Bollas A,etal. Nanopore sequencing technology, bioinformatics and applications[J]. Nature Biotechnology,2021,39(11):1348-1365.
[4] Kasianowicz JJ. Some Physics and Applications for DNA TransportThrough Single Nanopores. APS Meeting Abstracts, 2002.
[5] Butler TZ,Pavlenok M,Derrington IM, etal.Single-molecule DNA detection with an engineered MspA protein manopore[J].Proc Natl Acad Sci USA,2008,105(52):20647-20652.
[6] Manrao EA,Derrington IM,Laszlo AH,etal.Reading DAN at single-nucleotide resolution with a mutant MspA manoproe and phi29 DNA polymerase[J].Nat Biotechnol,2012,30(4):349-353.
[7] Zhou W Y, Tang D S , Yu-Bao Li , et al. Self-organized formation ofhexagonal Namopore arrays in anodic alumina [J]. Chinese Physics:English Version, 2001.
[8] Yuan Z,Wang C,Yi X,etal. Solid-statenanopore[J]. Nanoscale Res Lett,2018,13(1):56.
[9] 田李,张颖,赵云峰.新一代测序技术的发展和应用[J].生物技术通报,2015,31(11):1-8.
[10] 张小珍,尤崇革.下一代基因测序技术新进展[J]. 兰州大学学报(医学版),2016,42(3):73-80.
[11] 庄子,孟雨桐,刘润旸等.纳米孔测序技术及其在病原学诊断中的应用进展[J].江苏大学学报(医学版),2023,33(6):502-508
[12] 刘可君, 郭世富, 崔乐等.基因测序技术在临床检验领域的应用及国内外监管现状比较研究[J].中国药事,2018,32(11):1520-1530
审评六部 郝玮琳 供稿
(转载自国家药品监督管理局医疗器械技术审评中心网站)